Differential Diagnosis of COVID-19 and Community-acquired Pneumonia Using Different Machine Learning Methods
-
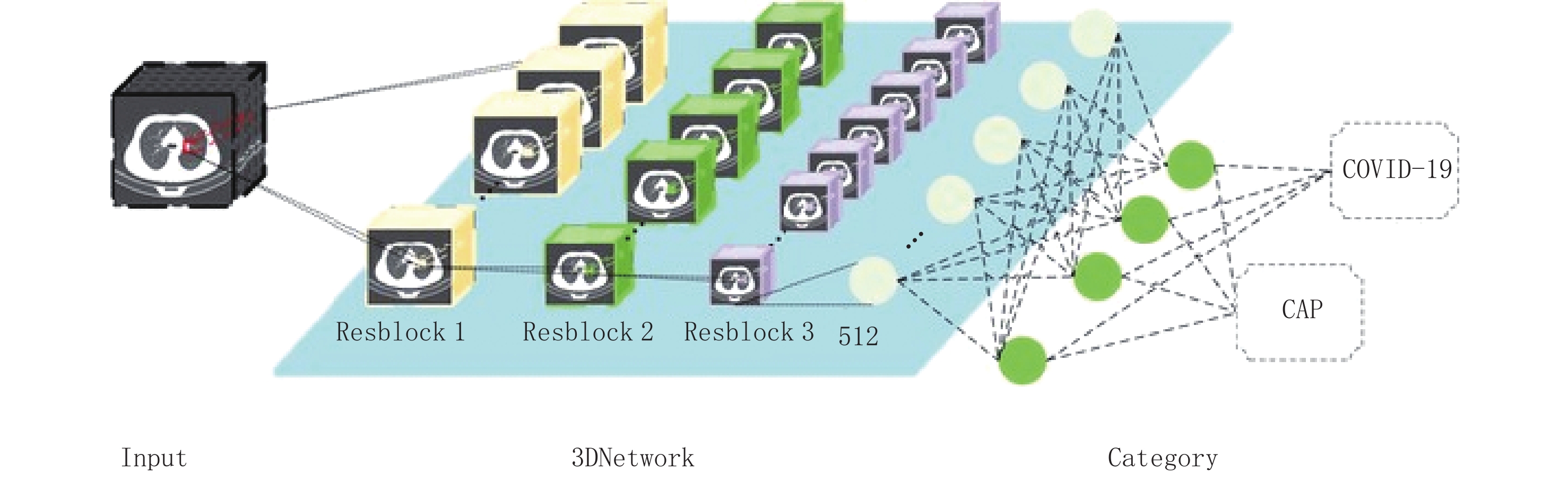
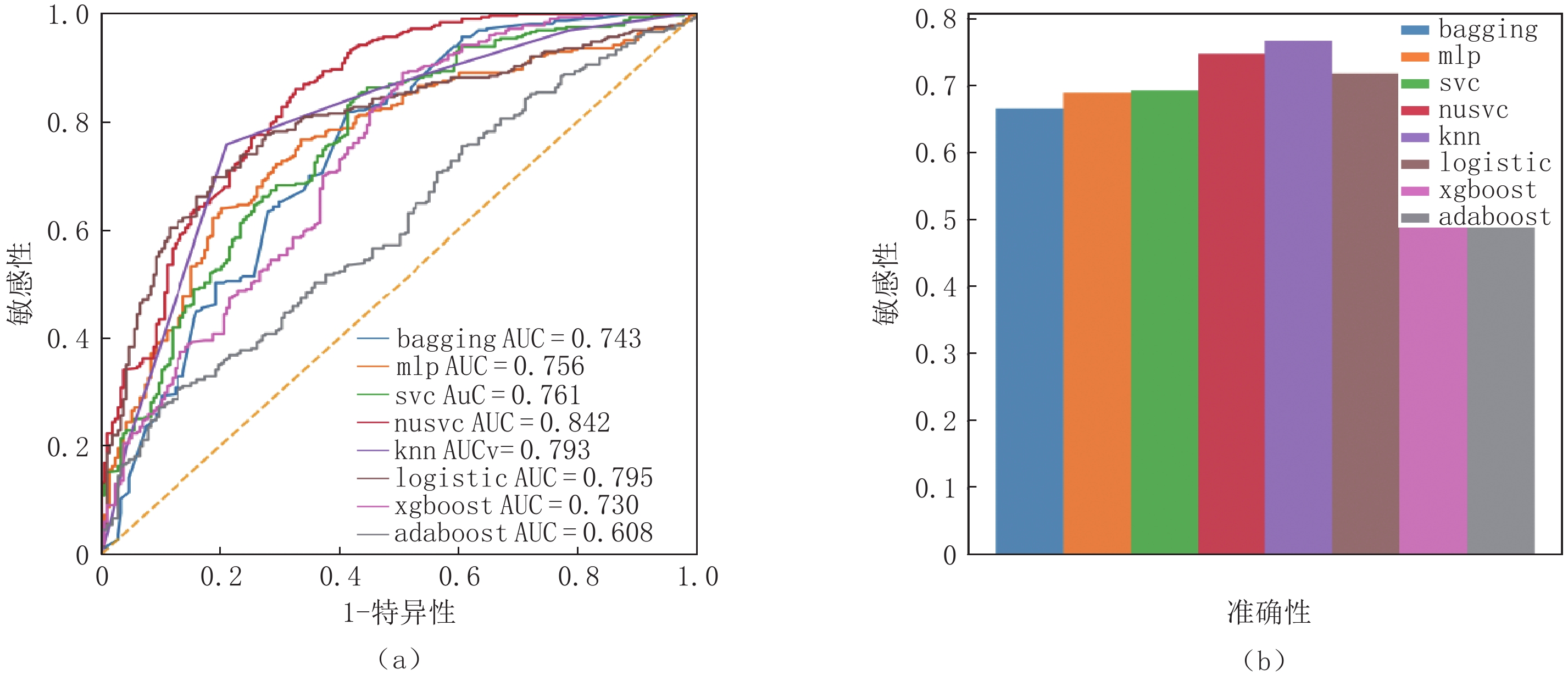
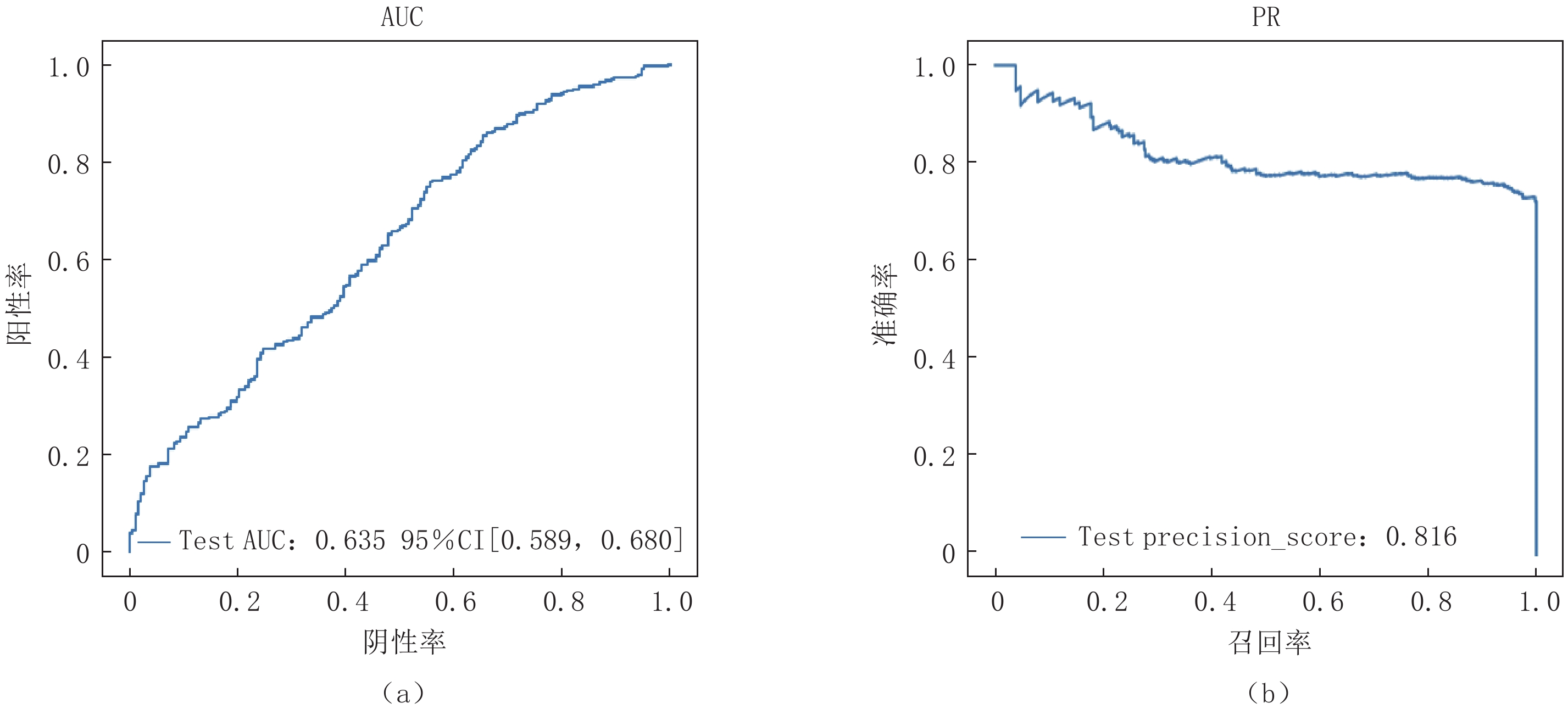
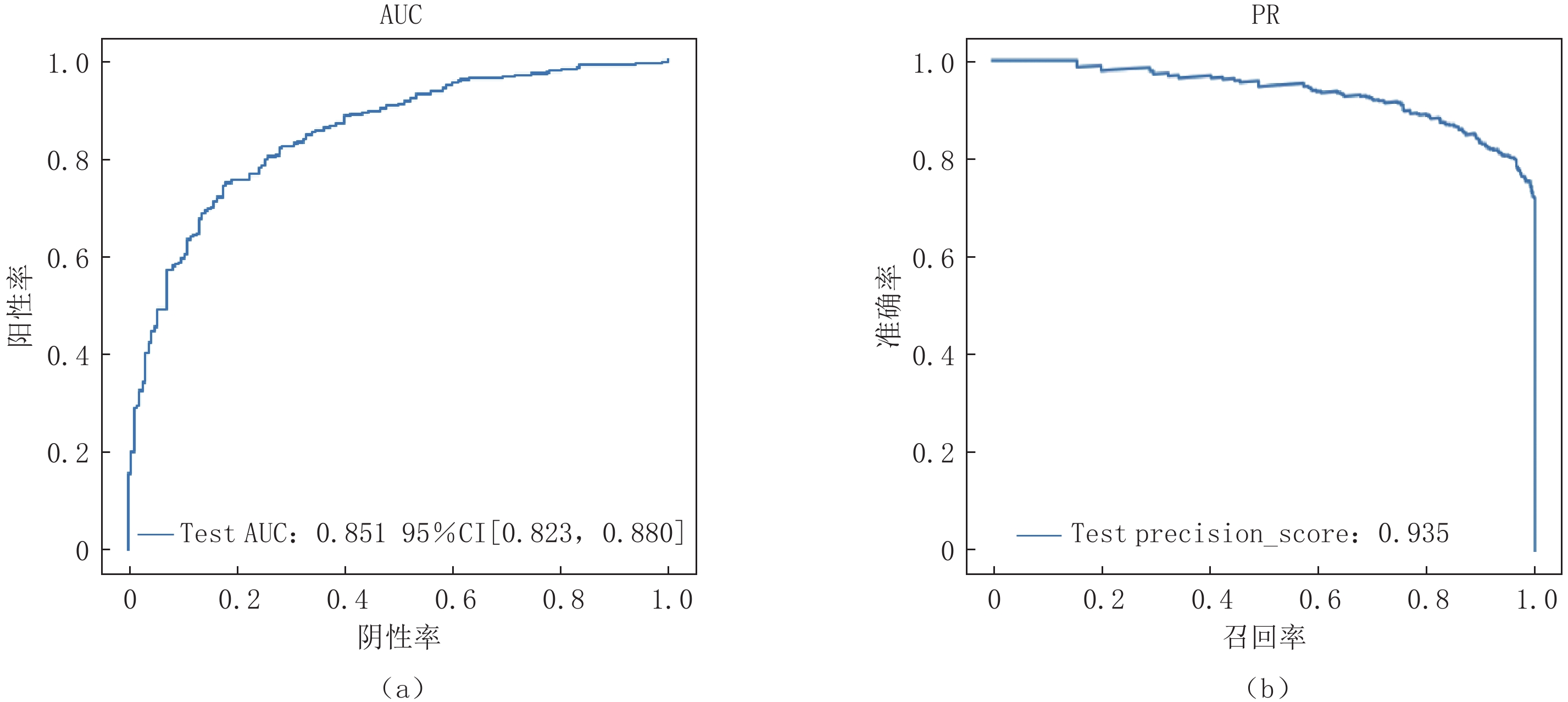
摘要: 目的:利用深度学习技术,全自动标注病变的计算机断层扫描(CT)数据,开发准确快速区分新型冠状病毒感染(COVID-19)和其他社区获得性肺炎的人工智能模型。方法:回顾性分析248例COVID-19患者及347例其他肺炎患者的资料,进行COVID-19与其他肺炎分类;在人工智能肺分割提取后将异常的CT图像特征降维,输入几种经典强化机器学习模型、三维卷积神经网络(3D CNN)和注意力多示例学习(Attention-MIL)深层神经网络架构中,模型诊断性能利用受试者工作特性(ROC)曲线、精确召回率(PR)曲线、曲线下面积(AUC)、敏感性、特异性、准确性指标进行评价。结果:在经典机器学习模型中K邻近算法(KNN)具有较好的效果,在外部测试集上的AUC值和平均精度(AP)值分别为0.79和0.89,平衡F分数(F1)值为0.76,准确率为0.75,敏感性为0.76,精确率为0.77;经典的3D CNN在外部测试集上效果良好,AUC值和AP值分别为0.64和0.82,F1值为0.71,准确率为0.78,敏感性为0.66,精确率为0.62;Attention-MIL模型在外部测试集上表现出更好的鲁棒性,AUC值和AP值分别为0.85和0.94,F1值达到0.82,准确率为0.92,敏感性为0.74,精确率为0.76。结论:与强化影像组学和3D CNN模型相比,深度学习Attention-MIL模型在鉴别诊断COVID-19和其他社区获得性肺炎上表现出更高的效能。
-
关键词:
- CT /
- Attention-MIL模型 /
- COVID-19 /
- 社区获得性肺炎
Abstract: Purpose: Utilizing deep learning techniques, this study aimed to develop an artificial intelligence model that automatically annotates lesion computed tomography (CT) data, accurately and rapidly distinguishing novel coronavirus pneumonia (COVID-19) from other community-acquired pneumonia cases. Methods: A retrospective analysis was conducted on data from 248 patients with COVID-19 and 347 patients with other types of pneumonia. The COVID-19 cases were differentiated from other pneumonia cases during classification. After performing artificial intelligence-based lung segmentation, the extracted abnormal CT image features were dimensionally reduced and inputted into various classical machine learning models, Three-dimensional convolutional neural network (3D CNN), and attention-Multiple-instance learning (MIL) deep neural network architectures. The diagnostic performance of the models was evaluated using metrics such as receiver operating characteristic (ROC) curves, Precision Recall (PR) curves, Area Under Curve (AUC), sensitivity, specificity, and accuracy. Results: Among the classical machine learning models, K-Nearest Neighbor (KNN)demonstrated good performance, with an AUC of 0.793, Average Precision (AP) of 0.886, Balanced F Score (F1-score) of 0.7608, accuracy of 0.7512, sensitivity of 0.7754, and precision of 0.7691 on the external test set. The classical 3D CNN model exhibited satisfactory performance on the external test set with an AUC of 0.635, AP of 0.816, F1-score of 0.7144, accuracy of 0.7783, sensitivity of 0.6603, and precision of 0.6200. The attention-MIL model showed better robustness on the external test set, achieving an AUC of 0.851, AP of 0.935, F1-score of 0.8193, accuracy of 0.9155, sensitivity of 0.7414, and precision of 0.7646. Conclusion: Compared to the radiomics-enhanced and 3D CNN models, the deep learning attention-MIL model exhibited better performance in the differential diagnosis of COVID-19 and other community-acquired pneumonia.-
Keywords:
- CT /
- attention-MIL model /
- COVID-19 /
- community-acquired pneumonia
-
近年来,人工智能(AI)技术在医学影像分析领域快速发展,在一份调查报告中显示,AI用于冠状动脉分析的普及率已从2019年占比6%迅速提高到了58%[1]。其中AI结合非门控肺CT扫描分析冠状动脉钙化已然成为了一个新兴研究方向[2−3],尤其是低剂量肺CT具有冠状动脉钙化检测和分级的潜力[4],并且表现出了可靠的重复性[5]。这种方法是在无心电门控监测下,利用常规肺部CT扫描结合AI测量钙化积分,从而大大提高了临床效率,并且为扩大筛查范围提供了重要保证。但是由于AI测量的准确性受到输入图像质量的影响[6],且常规肺部CT检查,扫描时间较长,往往存在心脏搏动伪影,这会影响冠状动脉的清晰度和AI测量钙化积分的准确性。因此,本研究探讨非门控大螺距肺CT联合AI技术,是否可以提高冠脉钙化积分的准确性以及在临床实践中的可行性。
1. 材料与方法
1.1 实验材料
Simens双源Force CT机(德国Simens公司);Simens Syngo.via工作站;联影AI门控钙化积分测量模块、联影AI非门控钙化积分测量模块。
1.2 一般资料
回顾性选取2022年7月至2022年11月,在我院同时进行非门控大螺距肺CT平扫和心电门控冠状动脉钙化积分CT扫描的患者30例。入组标准:未进行心脏搭桥手术;未安装心脏起搏器;未放置冠状动脉支架。排除标准:心电门控冠状动脉钙化积分为0;图像存在严重伪影影响诊断。最终纳入24例患者,平均年龄33 ~ 84(60.32±12.34)岁。男16例,女8例。BMI:20.4 ~ 38(25.98±3.74),心率46 ~ 97(66.42±10.96)次/分。
本研究获医学伦理委员会批准,伦理号为TREC2022-KY035。
1.3 扫描参数设置
1.3.1 非门控大螺距肺CT平扫参数
定位像:X射线投照角度选择0°(即前后位定位像),管电压120 kV,管电流20 mA。正式扫描:自动管电压调制技术(CarekV)和自动管电流调制技术(CareDose 4 D),参考管电压110 kV,参考管电流80 mAs,探测器宽度192×0.6 mm,旋转时间0.25 s,螺距3。
1.3.2 心电门控冠状动脉钙化积分CT参数
定位像扫描同上。正式扫描:CarekV,CareDose 4 D,参考管电压120 kV,参考管电流50 mAs,螺距0.15。采集R-R间期35%以及75%,选择冠状动脉显示好,伪影小的时相。其余参数与非门控大螺距肺CT扫描参数一致。
1.4 图像重组
两种扫描方式均重组心脏范围的轴位图像。滤过核:大螺距肺CT为BR40;冠状动脉钙化积分CT为QR36。其余参数均一致:窗宽/窗位345/50,层厚/间隔3 mm/1.5 mm。大螺距肺CT图像作为A组,冠状动脉钙化积分CT图像作为B组。
1.5 钙化积分测量及风险等级分级
A、B组图像分别采用3种方法测量AS:①Syngo.via工作站测量:将图像中CT值>130 HU的区域进行色彩标记,然后由图像后处理技师手动选择冠脉解剖结构后再自动计算得出AS。②AI测量:AI软件包括非门控钙化积分和门控钙化积分两个模块,分别用于测量A组和B组图像。两个模块均采用卷积神经网络的深度学习算法。③AI+手动校正测量:由图像后处理技师在AI测量的基础上,手动调整AI识别的冠状动脉钙化区域,包括增加或删除钙化区域。
Syngo.via工作站测量A、B组图像所得AS分别记录为A1组、B1组;AI非门控钙化积分模块测量A组图像所得AS为A2组,AI非门控钙化积分模块测量+手动校正A组图像所得AS为A3组;AI门控钙化积分模块测量B组图像所得AS为B2组,AI门控钙化积分模块测量+手动校正B组图像所得AS为B3组。对6组钙化积分进行风险分级,分级标准为:1级:小于1,为极低风险;2级:1 ~ 99,为低风险;3级:100 ~ 399,为中度风险;4级:大于400,为高风险。记录A1、A3以及B1、B3的所用时间。
1.6 辐射剂量评估
记录两种检查剂量报告上显示的容积剂量指数(CTDIvol)和剂量长度乘积(DLP)。再根据公式计算有效剂量(ED)。
$$ \mathrm{E}\mathrm{D}=\mathrm{D}\mathrm{L}\mathrm{P}\times 0.014 ^{[7]} $$ 1.7 统计分析
使用SPSS Statistics 27.0软件进行数据分析。连续变量正态分布用
$ \bar{{x}}\pm s $ 表示,非正态分布用M(P25,P75)形式表示。钙化积分作为连续变量,A1和B1组使用配对Wilcox检验评估是否存在统计学差异。A1、A2和A3组之间、B1、A2和A3组之间以及B1、B2和B3组之间分别使用多样本Friedman检验,P<0.05具有统计学差异。如有差异进一步行Nemenyi检验两两比较,P<0.017具有统计学差异(0.017为“Bonferroni校正”调整后的显著性阈值)。并且使用组内相关系数(interclass correlation coefficient,ICC)评估A1、B1组,A1、A2、A3组,B1、A2和A3组以及B1、B2、B3组之间的一致性。一致性较高>0.75;一致性较好0.40 ~ 0.75;一致性较差<0.40。钙化积分风险等级作为分类变量,使用加权Kappa系数来评估A1分别与B1、A2、A3组之间,B1分别与A2、A3、B2、B3组之间风险等级分级的一致性。一致性很强:0.8 ~ 1.0;一致性较强0.6 ~ 0.8;一致性中等0.4 ~ 0.6;一致性一般0.2 ~ 0.4;一致性较差<0.2。A1和A3之间及B1和B3之间所用时间使用配对Wilcox检验评估是否存在统计学差异。2. 结果
2.1 非门控大螺距肺CT与心电门控冠脉钙化积分CT扫描参数和辐射剂量统计(表1)
表 1 两种扫描方法扫描参数和辐射剂量Table 1. Scanning parameters and radiation doses in the two scanning methods扫描方法 实际管电压(kV) 实际管电流(mAs) 容积剂量指数(mGy) 剂量长度乘积(mGy·cm) 有效剂量(mSv) 非门控大螺距肺CT 120(4)* 84-102(4)* 4.0±0.7 149.2±31.2 2.1±0.4 110(13)* 72-104(13)* 100(7)* 102-158(7)* 心电门控冠状动脉
钙化积分CT120(24)* 22-64(24)* 9.0±2.8 152.2±49.2 2.1±0.7 注:*括号内数值代表病例数。 2.2 非门控大螺距肺CT与心电门控冠脉钙化积分CT图像分别使用工作站和AI测量所得钙化积分(表2,图1)
表 2 两种扫描方式图像使用3种测量方法所得钙化积分统计分析Table 2. Calcification scores of the two scanning images using three measurement methods扫描方式 测量方法 Syngo.via
工作站测量AI测量 AI+手动
校正测量X2 P ICC 工作站与AI
测量Kappa工作站与AI+
手动校正
测量Kappa非门控大螺
距肺CT99.70(32.20,374.80) 97.00(31.00,337.00)1 97.50(31.00,351.25)1 27.163 <0.001 0.988
(0.977,0.995)*0.818 0.818 心电门控冠
脉钙化积分CT96.75(30.90,363.40) 90.97(38.59,329.48)2 95.29(30.52,356.43)2 10.900 0.004 0.980
(0.960,0.990)*1.000 1.000 注:1使用AI非门控钙化积分模块测量;2使用AI门控钙化积分模块测量;ICC,组内相关系数;*括号内数值为95%置信空间;Kappa,加权Kappa系数。 2.3 统计分析结果
大螺距肺CT的3种测量方法所得AS有统计学差异,见表2(X2=27.163,P<0.001),对A1、A2和A3组之间使用Nemenyi检验进一步两两比较得出:A1组分别与A2、A3组存在统计学差异(P=0.001、0.011,均<0.017),A2与A3组之间无统计学差异(P=0.107>0.017),A2与A3之间手动校正不为0的共14例,占58%。但AS一致性较高(ICC:0.988)。
心电门控冠脉钙化积分CT使用3种方法测得AS使用多样本Friedman检验时,P=0.004<0.05(表2)。对B1、B2和B3组之间使用Nemenyi检验进一步两两比较得出:B1与B2、B1与B3、B2与B3均无统计学差异(P=0.017、0.900、0.038,均P≥0.017)。因两两比较不存在统计学差异,因此门控冠脉钙化积分CT使用3种方法测得AS不存在统计学差异。B2与B3之间手动校正不为0的共16例,占67%。
大螺距肺CT联合AI(X2=4.795,P=0.091、ICC:0.990)、大螺距肺CT工作站(Z=0.912,P=0.362、ICC:0.988)、门控钙化积分联合AI(X2=10.900,P=0.004、ICC:0.980)分别与门控钙化积分工作站测得AS,均无统计学差异且一致性较高。
非门控大螺距肺CT联合AI及AI+手动校正测量,与门控钙化积分工作站测得AS之间一致性ICC为0.990(0.981,0.995),风险分级Kappa系数:A2与B1、A3与B1之间均为0.925。
A1组与A3组所用时间分别为97(31,337)秒、12.5(5,16.3)秒,存在统计学差异(Z =4.200,P<0.001)。B1组与B3组所用时间分别为32.5(17.3,51.3)秒、15(5,20)秒,存在统计学差异(Z =4.049,P<0.001)。
3. 讨论
随着冠状动脉疾病(CAD)在全球范围内的流行率不断上升,早期诊断和风险评估变得尤为重要。冠状动脉钙化积分(CAC)是评估冠状动脉疾病风险的一个重要指标,已被证明与心血管事件的风险密切相关。非门控肺CT作为一种快速且辐射剂量较低的成像技术,为冠状动脉钙化评估提供了新的方法[8−9],加上AI算法的引入,则进一步提高了CAC评分的自动化和准确性[9−10],为筛查心脏状况提供了一个有价值的工具。常规肺平扫CT[8]及低剂量肺CT[10]已经被证实可以较为准确地测量钙化积分,却仍存在噪声大或存在运动伪影的问题,有时对风险等级产生较大影响。本文通过大螺距联合自动调制技术,在提高时间分辨力、提升图像质量、优化患者辐射剂量(仅约4.0 mGy)的同时,进一步提高钙化积分准确性和风险分级的一致性。
研究结果显示:大螺距肺CT与冠脉钙化积分CT图像使用工作站测量时,钙化积分不存在显著差异,且一致性高达98.8%。相较Budoff[11]与Yu[2]等人研究结果一致性94%和86%,本研究具有更高的一致性。究其原因是大螺距肺CT图像较低剂量肺CT图像质量好,噪声水平低,使得钙化积分测量结果更准确。顾耕[12]等人研究显示,非门控大螺距肺CT平扫与心电门控钙化积分CT所得钙化积分结果有显著差异,并且大螺距肺CT钙化积分值低于门控钙化积分,与本研究结果相反。分析原因,钙化积分CT扫描时采用的前瞻性门控的单一时相(R-R间期75%),而本研究采用了R-R间期35%及75%双时相采集,虽然辐射剂量有所增加,但是可以重建出图像伪影小,冠脉显示更清晰的时相,因此得到了两者无显著差异的结果。此外,本研究中大螺距肺CT图像采集过程中,采用的自动调制技术,实际管电压值并非全为120 kV,约80%患者为100和110 kV,随管电压降低CT值增加,因此大螺距肺CT较门控钙化积分CT所测得的AS高一些,因其影响了钙化积分阈值的测量。但是此变化未对两种采集方式所得AS的差异性(P=0.362)和一致性(ICC:0.988)产生影响,其中仅有3例患者风险分级发生变化,相较门控钙化积分CT的风险分级高出一个等级,但一致性高达0.818。因此可以证明非门控大螺距肺CT代替门控冠状动脉钙化积分CT用于钙化积分评估的可行性。
大螺距肺CT平扫联合AI非门控钙化积分测量模块,通过对大量肺CT图像的反复训练,卷积神经网络的不断学习,建立肺CT测量钙化积分的优质模型。但是研究发现,无论是AI自动或是AI自动+手动校正测量,所得AS与工作站测量都存在差异,且AS出现降低的情况。但大螺距肺CT平扫联合AI与门控钙化积分工作站测量结果相较,并不存在统计学差异。大螺距肺CT图像经过AI训练后所得的AS反倒与门控冠脉钙化积分的工作站测量值更接近。究其原因,可能是AI非门控钙化积分模块在模型训练过程中,以肺CT图像作为输入,以门控钙化积分图像作为预测目标,通过前向传播计算预测结果,然后通过反向传播调整模型参数,因此可以得到与“金标准”(心电门控钙化积分工作站测量结果)更为相近的结果。并且AS一致性高达0.990,风险分级Kappa系数一致性为0.925,以及较工作站测量所用时间显著减少,效率更高。因此大螺距肺CT平扫联合AI保有较高的风险等级诊断效能及临床筛查价值,这与樊荣荣[13]、闫玉辰[14]以及 Andre F[15]等人的研究结论一致。
此外,本研究中心电门控钙化积分CT联合AI,可得到与工作站无差异的结果,且风险分级一致性高达1.000。AS在经过AI+手动校正后的测量结果具有更高的准确性,与陈丽虹[16]与Covas P[17]等人的研究结果一致。
综上所述,非门控大螺距肺CT图像可以用于钙化积分测量,使用AI评估具有较高的准确性,同时降低筛查的辐射剂量,节约检查和诊断时间,可以做到无人为干扰的结果统计,因此具有较高的临床应用价值。而且非门控大螺距肺CT可以使患者少接受冠脉钙化积分扫描中约2.1 mSv的有效剂量、以及约9.0 mGy左右的CTDIvol,尤其针对女性患者,可以大大减低局部辐射剂量,对于扫描范围内的辐射敏感器官-乳腺起到了很好的保护作用。通过一种CT检查,在一次扫描中获取更多的诊疗信息,对心血管疾病的大范围筛查和早诊早治提供了有力保障。
本研究存在的不足之处:(1)样本量较小,无法根据患者自身特征进行分组讨论。(2)本研究所使用成像设备与AI软件源于不同公司,且较为单一,可能会对结果的泛化产生影响。
-
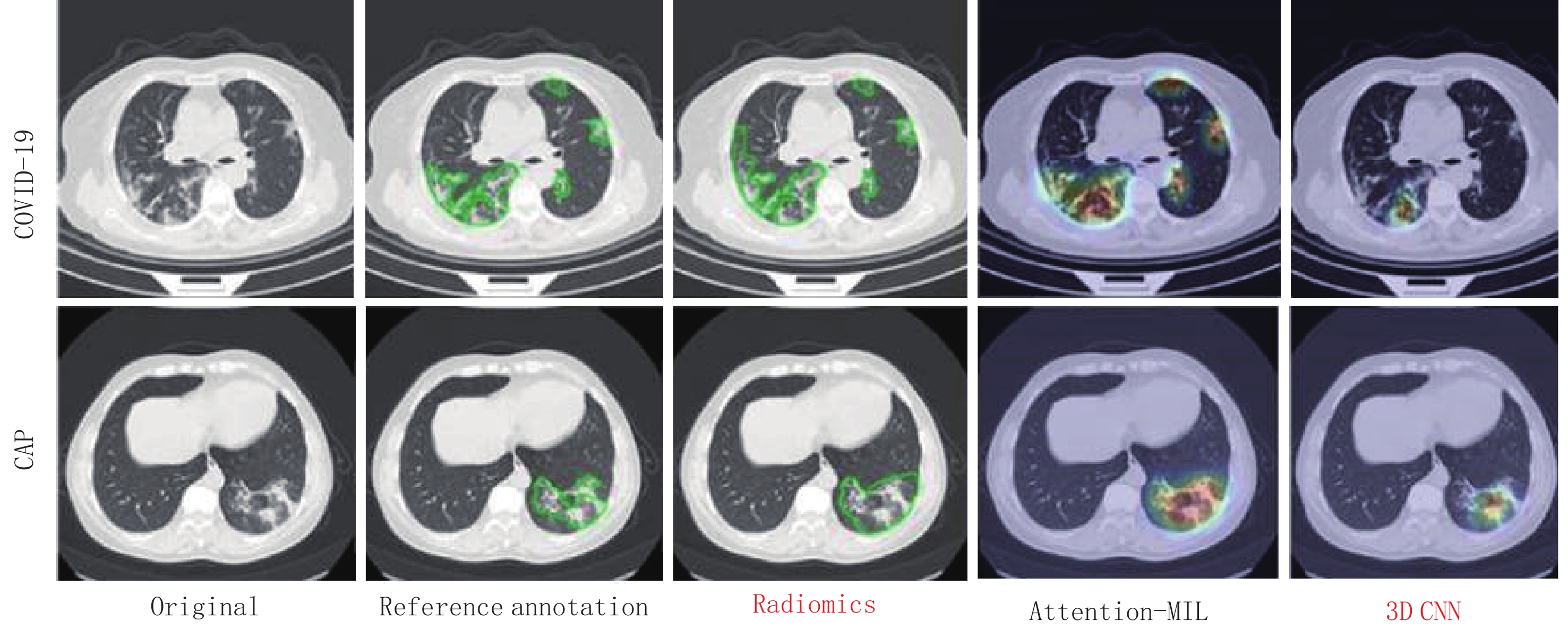
图 6 新型冠状病毒感染、社区获得性肺炎者使用梯度加权激活映射或Grad-CAM方法生成的注意力热图
热图是标准的Jet颜色图,并与原始图像重叠。红色突出显示与预测类别关联的激活区域。
Figure 6. Coronavirus disease 2019 (COVID-19), a representative example of attention heatmaps generated with data from individuals with community-acquired pneumonia using gradient-weighted category activation mapping or the Grad-CAM method-pneumonia
表 1 不同医院患者的统计数据汇总
Table 1 Summary of the statistical data of patients from different hospitals
不同医院肺炎患者 病例数(CT数)/例 年龄/岁 男/例 女/例 内蒙古人民医院 COVID-19 80 45±13.11 34~77 36 44 CAP 102 56±14.12 45~67 57 45 金门县人民医院 COVID-19 143(143) 44.95±15.12 2~86 73 70 浙江省人民医院 COVID-19 4(4) 43±13.13 26~59 1 3 CAP 35(35) 42.08±14.95 10~66 21 14 浙江大学医学院附属邵逸夫医院 COVID-19 8(8) 42.75±6.33 33~51 4 4 CAP 210(334) 44.05±16.77 15~85 103 107 台州市中心医院 COVID-19 13(13) 47.76±14.22 31~74 6 7 表 2 各种方法在外部测试集上的表现评价指标
Table 2 Performance evaluation indicators for each method on independent test sets
测试集 F1值 准确率/% 召回率/% 精确率/% Adaboost 0.55 0.56 0.55 0.55 bagging 0.66 0.65 0.68 0.67 KNN 0.76 0.75 0.77 0.77 logistic 0.72 0.75 0.74 0.72 MLP 0.69 0.69 0.71 0.69 nusvc 0.74 0.75 0.76 0.75 SVC 0.68 0.69 0.68 0.69 xgboost 0.60 0.60 0.62 0.59 表 3 不同机器学习框架在COVID-19独立测试集上的性能
Table 3 Performance of different machine learning frameworks on COVID-19 independent test sets
Group/COVID-19 敏感性/% 特异性/% AUC P KNN 77 67 73 P<0.001 3D CNN 78 69 76 P<0.001 Attention-MIL 90 96 85 P<0.001 -
[1] CHEN N, ZHOU M, DONG X, et al. Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: A descriptive study[J]. Lancet, 2020, 395(10223): 507−513. DOI: 10.1016/S0140-6736(20)30211-7.
[2] GAO Y, YAN L, HUANG Y, et al. Structure of the RNA-dependent RNA polymerase from COVID-19 virus[J]. Science, 2020, 368(6492): 779−782. DOI: 10.1126/science.abb7498.
[3] LI Q, GUAN X, WU P, et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia[J]. New England Journal of Medicine, 2020, 382(13): 1199−1207. DOI: 10.1056/NEJMoa2001316.
[4] HOLSHUE M L, DEBOLT C, LINDQUIST S, et al. First case of 2019 novel coronavirus in the United States[J]. New England Journal of Medicine, 2020, 382(10): 929−936. DOI: 10.1056/NEJMoa2001191.
[5] AI T, YANG Z, HOU H, et al. Correlation of chest CT and RT-PCR testing in coronavirus disease 2019 (COVID-19) in China: A report of 1014 cases[J]. Radiology, 2020, 296(2): E32−E40. DOI: 10.1148/radiol.2020200642.
[6] FANG Y, ZHANG H, XIE J, et al. Sensitivity of chest CT for COVID-19: Comparison to RT-PCR[J]. Radiology, 2020, 296(2): E115−E117. DOI: 10.1148/radiol.2020200432.
[7] 刘玉建, 仲建全, 冯浩, 等. 新型冠状病毒肺炎患者的高分辨率 CT 影像学特征[J]. 医疗装备, 2022,35(11): 1−4. LIU Y J, ZHONG J Q, FENG H, et al. Imaging characteristics of high resolution CT for patients with corona virus disease 2019[J]. Medical Equipment, 2022, 35(11): 1−4. (in Chinese).
[8] HUANG C, WANG Y, LI X, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China[J]. Lancet, 2020, 395(10223): 497−506. DOI: 10.1016/S0140-6736(20)30183-5.
[9] MEI X, LEE H C, DIAO K Y, et al. Artificial intelligence-enabled rapid diagnosis of patients with COVID-19[J]. Nature Medicine, 2020, 26(8): 1224−1228. DOI: 10.1038/s41591-020-0931-3.
[10] CHEN Y, FAN S, CHEN Y, et al. Vessel segmentation from volumetric images: A multi-scale double-pathway network with class-balanced loss at the voxel level[J]. Medical Physics, 2021, 48(7): 3804−3814. DOI: 10.1002/mp.14934.
[11] YE H, GAO F, YIN Y, et al. Precise diagnosis of intracranial hemorrhage and subtypes using a three-dimensional joint convolutional and recurrent neural network[J]. European Radiology, 2019, 29(11): 6191−6201. DOI: 10.1007/s00330-019-06163-2.
[12] KERMANG D S, GOLDBAUM M, CAI W, et al. Identifying medical diagnoses and treatable diseases by image-based deep learning[J]. Cell, 2018, 172(5): 1122−1131.e9. DOI: 10.1016/j.cell.2018.02.010.
[13] RAJARAMAN S, CANDEMIR S, KIM I, et al. Visualization and interpretation of convolutional neural network predictions in detecting pneumonia in pediatric chest radiographs[J]. Applied Sciences-Basel, 2018, 8(10): 1715. DOI: 10.3390/app8101715.
[14] WYNANTS L, Van CALSTER B, COLLINS G S, et al. Prediction models for diagnosis and prognosis of COVID-19 infection: Systematic review and critical appraisal[J]. British Medical Journal, 2020, 369: m1328. DOI: 10.1136/bmj.m1328.
[15] ZHANG X, WANG D, SHAO J, et al. A deep learning integrated radiomics model for identification of coronavirus disease 2019 using computed tomography[J]. Scientific Reports, 2021, 11(1): 3938. DOI: 10.1038/s41598-021-83237-6.
[16] HUANG Y Q, LIANG C H, HE L. Preoperative prediction of lymph node metastasis in colorectal cancer[J]. Journal of Clinical Oncology, 2016, 34(18): 2157−64. DOI: 10.1200/JCO.2015.65.9128.
[17] PARMAR C, GROSSMANN P, BUSSINK J, et al. Machine learning methods for quantitative radiomic biomarkers[J]. Scientific Reports, 2015, 15: 13087. DOI: 10.1038/srep13087.
[18] NIETHAMMER M, KWITT R, VIALARD F X. Metric learning for image registration[J]. Proc EEE Computer Society Conference on Computer Vision and Pattern Recognition, 2019: 8455-8464. DOI: 10.1109/cvpr.2019.00866.
[19] ZHANG X, LU D, GAO P, et al. Survival-relevant high-risk subregion identification for glioblastoma patients: The MRI-based multiple instance learning approach[J]. European Radiology, 2020, 30(10): 5602−5610. DOI: 10.1007/s00330-020-06912-8.
[20] LIU Y, FU Q, PENG X, et al. Attention-based deep multiple-instance learning for classifying circular RNA and other long non-coding RNA[J]. Genes (Basel), 2021, 12(12): 2018. DOI: 10.3390/genes12122018.
[21] DELONG E R, DELOONG D M, CLARKE-PEARSON D L. Comparing the areas under two or more correlated receiver operating characteristic curves: A nonparametric approach[J]. Biometrics, 1988, 44(3): 837−845. doi: 10.2307/2531595
[22] SELVARAJU R R, COGSWELL M, DAS A, et al. Grad-cam: Visual explanations from deep networks via gradient-based localization[J]. Proceedings of the IEEE International Conference on Computer Vision, 2017. DOI: 10.1109/ICCV.2017.74.
[23] MARTIN J, TENA N, ASUERO A G. Current state of diagnostic, screening and surveillance testing methods for COVID-19 from an analytical chemistry point of view[J]. Microchemical Journal, 2021, 167: 106305. DOI: 10.1016/j.microc.2021.106305.
[24] XU X, JIANG X, MA C, et al. A deep learning system to screen novel coronavirus disease 2019 pneumonia[J]. Engineering (Beijing), 2020, 6(10): 1122−1129. DOI: 10.1016/j.eng.2020.04.010.
[25] ABBAS A, ABDELSAMEA M, GABER M. Classification of covid-19 in chest X-ray images using DeTraC deep convolutional neural network[J]. Applied Intelligence, 2021, 51(2): 854−864. DOI: 10.1007/s10489-020-01829-7.
[26] GOZES O, FRID-ADAR M, SAGIE N, et al. Detection and analysis of COVID-19 in medical images using deep learning techniques[J]. Scientific Reports, 2021, 11(1): 19638. DOI: 10.1038/s41598-021-99015-3.
[27] CHEN J, WU L, ZHANG J, et al. Deep learning-based model for detecting 2019 novel coronavirus pneumonia on high-resolution computed tomography: A prospective study[J]. Scientific Reports, 2020, 10(1): 19196. DOI: 10.1038/s41598-020-76282-0.
[28] WANG S, KANG B, MA J, et al. A deep learning algorithm using CT images to screen for corona virus disease (COVID-19)[J]. European Radiology, 2021, 31(8): 6096-6104.
[29] LI Z, ZHONG Z, LI Y, et al. From community-acquired pneumonia to COVID-19: A deep learning-based method for quantitative analysis of COVID-19 on thick-section CT scans[J]. European Radiology, 2020, 30(12): 6828−6837. DOI: 10.1007/s00330-020-07042-x.
[30] CHOUAT I, ECHTIOUI A, KHEMAKHEM R, et al. COVID-19 detection in CT and CXR images using deep learning models[J]. Biogerontology, 2022, 23(1): 65−84. DOI: 10.1007/s10522-021-09946-7.





 下载:
下载: